功能蛋白质组学实验室
导师介绍

廖鲁剑 教授
2002年12月在美国Bowling Green州立大学获得计算机科学硕士和生物科学/神经生物学博士学位,之后在美国Emory大学及Scripps 研究所从事博士后研究,后以Staff Scientist任职于加州Scripps 研究所John Yates实验室。2012年5月回国任中科院上海生化与细胞生物研究所/国家蛋白质科学研究中心研究员,并获中科院上海生科院特殊人才计划支持。2013年8月受聘华东师范大学生命医学研究所担任生物医学教授、博士生导师,组建功能蛋白质组学实验室。主要研究方向为利用蛋白质组学方法结合分子、细胞、动物等手段研究细胞器质量控制在神经退行性疾病发生中的作用。在确定神经退行性疾病主要病变的蛋白组成和定量蛋白质组学新方法探索做出开拓性的工作,成果发表于JBC,J Proteome Res, PNAS,Nature Methods。在SCI影响因子>5的各类专业期刊上发表第一或共同第一作者研究论文10篇,应邀综述1篇。利用高精度定量质谱的合作研究发现重要疾病模型的蛋白质差异表达和蛋白修饰,研究成果发表于Immunity,Cell,PNAS,Science,Nat.Immuology,EMBO J.,Nat.Cell Biol.等杂志。
主要研究方向
在后基因组时代,系统研究细胞或机体的蛋白质在时间,空间水平的定量变化,翻译后修饰,以及蛋白复合体的动态变化,已经在许多方向为进一步理解生命过程的基本原理,深入了解重大疾病的发生发展机理开辟了新途径。基于液相色谱-高分辨质谱联用的蛋白组学在这些研究方向起着举足轻重的作用。本实验室利用先进的质谱技术,结合分子生物学、细胞生物学、生物化学何动物模型等多种研究手段,探索蛋白质及细胞器稳态在神经退行性疾病发病过程中的作用。尤其关注蛋白激酶和泛素连接酶的功能变化,以及线粒体和内质网质量控制系统的异常导致神经细胞退行性变的机理。
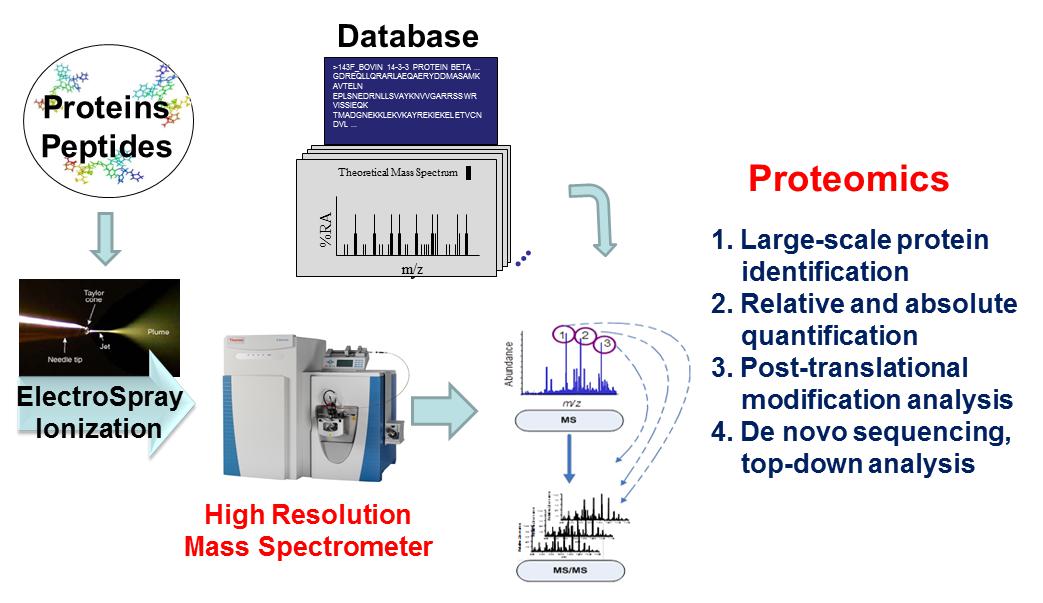
蛋白质组学技术开发
基于液相色谱-高分辨质谱的蛋白质组学已经在过去十年有了长足的发展。在技术上,蛋白质组学能在一次实验中常规鉴定出五-六千多个蛋白质。经过特殊的实验设计和样品处理,蛋白质组学能对不同的生物样品中蛋白质差异表达进行分析,也能大规模检测和定量蛋白质组的翻译后修饰。在蛋白质组学技术开发领域,我们主要关注以下问题:
大规模、深度覆盖地鉴定和定量磷酸化蛋白质组
利用定量质谱研究蛋白质-蛋白质之间相互作用
建立筛查在神经退行性疾病中起重要作用的蛋白酶底物的化学生物学方法
蛋白质磷酸化和其它修饰在神经退行性疾病发生中的作用
神经退行性疾病,包括老年性痴呆(Alzheimer’s),帕金森氏病(Parkinson’s), 肌萎缩性脊髓侧索硬化症(ALS)等,已经在有家族史的病人中找到多种基因突变。其中很多基因编码的蛋白质在神经系统的信号传导中起到举足轻重的作用,其中包括蛋白激酶,泛素连接酶和水解酶。我们将利用真核细胞或大小鼠原代培养的神经细胞表达这些基因,利用定量质谱技术寻找跟这些蛋白相互作用的蛋白复合体,并且搜寻这些激酶和泛素连接酶的底物。
利用定量磷酸化蛋白质组学方法(TMT)寻找蛋白PINK1激酶的底物和下游信号分子,已经这些底物在保护神经细胞免受氧化损伤的机制
利用定量蛋白质组学方法(SILAC)研究自噬蛋白缺少突变导致细胞器质量控制失常及神经退行性变的机理
研究团队

研究骨干:廖鲁剑教授,唐彬 工程师
技术骨干:方后琴
在读博士研究生:余春雷,王婷婷,万慧达,毛琳,曾秋芳,张云鹏,徐越明
在读硕士研究生:王琪,王靖宇,赵宛冰,刁苗苗,闫凯
代表性论文 (# 共同第一作者,* 通讯作者)
Wan H, Tang B*, Liao X, Zeng Q, Zhang Z, Liao L*. Analysis of neuronal phosphoproteome reveals PINK1 regulation of BAD function and cell death. Cell Death & Diff. 2017, Accepted
Tan GH, Liu YY, Wang L, et al., Liao LJ, Wu ZY, Xiong ZQ. PRRT2 deficiency induces paroxysmal kinesigenic dyskinesia by regulating synaptic transmission in cerebellum. Cell Res. 2017, doi: 10.1038/cr.2017.128. PMID: 29056747
Wang T, Yu Q, Li J, Hu B, Zhao Q, Ma C, Huang W, Zhuo L, Fang H, Liao L, Eugene Chin Y, Jiang Y. O-GlcNAcylation of fumarase maintains tumour growth under glucose deficiency. Nat Cell Biol. 2017, 19(7):833-843. PMID:28628081
Wei W, Mao A, Tang B, Zeng Q, Gao S, Liu X, Lu L, Li W, Du JX, Li J, Wong J*, Liao L*. Large-Scale Identification of Protein Crotonylation Reveals Its Role in Multiple Cellular Functions. J Proteome Res. 2017, 16(4):1743-1752.PMID:28234478
Xu DQ, Wang Z, Wang CY, et al., Liao LJ, Chen Y. PAQR3 controls autophagy by integrating AMPK signaling to enhance ATG14L-associated PI3K activity. EMBO J. 2016, 35(5):496-514. PMID:26834238
Park Y, Jin HS, Lopez J, Lee J, Liao L, Elly C, Liu YC. SHARPIN controls regulatory T cells by negatively modulating the T cell antigen receptor complex. Nat Immunol. 2016 Mar;17(3):286-96. PMID:26829767
Tang B, Wang T, Wan H, Han L, Qin X, Zhang Y, Wang J, Yu C, Berton F, Francesconi W, Yates JR 3rd, Vanderklish PW, Liao L*. Fmr1 deficiency promotes age-dependent alterations in the cortical synaptic proteome. Proc Natl Acad Sci U S A. 2015, 112(34):E4697-706. PMID:26307763
Qin X, Zheng C, Yates JR 3rd, Liao L*. Quantitative phosphoproteomic profiling of PINK1-deficient cells identifies phosphorylation changes in nuclear proteins. Mol Biosyst. 2014, 10(7):1719-29. PMID:24626860
Li K, Zhou T, Liao L, Yang Z, Wong C, Henn F, Malinow R, Yates JR, Hu H. βCaMKII in Lateral Habenula Mediates Core Symptoms of Depression. Science 2013;341:1016-1020.
Jin HS, Liao L, Park Y, Liu YC. Neddylation pathway regulates T-cell function by targeting an adaptor protein Shc and a protein kinase Erk signaling. Proc Natl Acad Sci U S A. 2013;110(2):624-9.
Sando R 3rd, Gounko N, Pieraut S, Liao L, Yates JR 3rd, and Maximov A. HDAC4 Governs Transcriptional Programs Essential for Synaptic Development and Function. Cell. 2012; 151(4):821-34.
Liao L, Sando RC, Farnum JB, Vanderklish PW, Maximov A, and Yates JR 3rd. 15N labeled brain enable quantification of proteome and phosphoproteome in neurons. J Proteome Res. 2012; 11(2):1341-53
Hart JR, Liao L, Yates JR 3rd, Vogt PK. An essential role of signal transducer and activator of transcription 3 in phosphatidylinositol 3-kinase-induced oncogenic transformation. Proc Natl Acad Sci USA. 2011; 108(32):13247-52
Hart JR*, Liao L*, Ueno L, Yates JR 3rd, Vogt PK. Protein expression profiles of C3H 10T1/2 murine fibroblasts and of isogenic cells transformed by the H1047R mutant of phosphoinositide 3-kinase (PI3K). Cell Cycle. 2011; 10(6):971-6
Huang H#, Jeon M#, Liao L#, Yang C, Elly C, Yates JR, and Liu YC. K33-Linked Polyubiquitination of T Cell Receptor-ζ Regulates Proteolysis-Independent T Cell Signaling. Immunity. 2010; 33(1):60-70 (# Co-first author)
Liao L#, McClatchy DB#, Yates JR. Primer: shotgun proteomics in neuroscience. (Review) Neuron. 2009; 63(1):12-26 (# Co-first author)
Park SK#, Liao L#, Kim JY, and Yates JR. A computational approach to correct arginine-to-proline conversion in quantitative proteomics. Nature Methods. 2009; 6(3):184-185 (# Co-first author)
Liao L, Park SK, Xu T, Vanderklish PW, and Yates JR. Quantitative proteomic analysis of primary neurons reveals diverse changes in synaptic protein content in fmr1 knockout mice. Proc Natl Acad Sci USA. 2008; 105(40):15281-6
Liao L#, McClatchy DB#, Park SK, Xu T, Lu B and Yates JR. Quantitative analysis of brain nuclear phosphoproteins identifies developmentally regulated phosphorylation events. J Proteome Res. 2008; 7(11):4743-55 (# Co-first author)
Xia Q#, Liao L#, Cheng D, Duong D, Gearing M, Lah JJ, Levey AI, and Peng J. Identification of novel kinases and ubiquitin ligases associated with Lewy bodies by comparative proteomics. Frontiers in Bioscience. 2008; 13:3850-6 (# Co-first author).
Liao L, Cheng D, Wang J, Duong DM, Losik TG, Gearing M, Rees HD, Lah JJ, Levey AI, Peng J. Proteomic characterization of postmortem amyloid plaques isolated by laser capture microdissection. J Biol Chem. 2004; 279(35):37061-8.


