生物信息学实验室
 | 石铁流 教授 于1992年在中科院上海植物生理研究所(现中科院上海植物生理生态研究所)获得植物生理专业硕士学位。1993年赴美留学,就读于Louisville大学,攻读分子生物学博士学位。在读期间于1999年获得计算机硕士学位,2000年初通过答辩,获得分子生物学专业的哲学博士学位。2002年5月回国,加入到中科院上海生命科学研究院生物信息中心。2008年底加入到华东师范大学,为生命科学学院和上海市调控生物学重点实验室特聘教授。 回国这些年来,共发表了SCI收录的文章40多篇。先后特邀为Plant Cell,Plant Physiology,Bioinformatics,BMC系列期刊审稿。
|
学术团队
研究骨干
石铁流 教授
博士研究生
赵琛,李鹏,崔健,吕清,陈赓
硕士研究生
胡鹏展,刘艳丽,卢冰心,方钊,薛瑞超,丁姗姗,涂海波,史彩萍,杨健民,屈雄飞,陈龙,周培颍,陈吉伟,杜宇,吉翔骏,冯普文,王欢,曹瑞芳,汪皓,杨娟,张立,张冠廷

一、蛋白质相互作用
主要是利用生物信息学的方法和手段对数据进行整合,并在此基础上对不同物种全基因组进行蛋白质相互作用的预测,并根据高通量数据进行动态的蛋白质相互作用网络的构建和分析。
二、基因调控网络
基因调控网络是开发预测基因调控网络的新方法,包括芯片数据分析分析方法(差异表达基因的挑选,生物途径对接和分析等),结合整合现有的转录因子结合位点的实验数据,构建生命活动中的动态基因调控网络,并探索新的转录调控机制。
三、疾病新基因及新的分子标记发现
对已有的研究文献进行数据挖掘,收集和整理已发现的疾病基因,建立相关的数据库应用系统,并利用我们自己开发的算法对数据进行深入的分析,发现新的与疾病可能相关的基因,为研究所其它实验室提供重要的线索。同时,我们基于基因表达谱,开发不同的算法来提取与疾病相关的特征基因,用于疾病的临床诊断和预后评估。
四、药物相互作用
基于基因表达谱和化合物分子结构的分析,利用已知化合物的作用表达谱及机理来研究未知化合物的靶点和机理。同时,复合用药是下一代新药开发的主要方向之一,我们正在利用生物信息学的手段开发相关的算法来研究不同药物间的相互作用。
五、进行高通量数据的分析,为其他实验室提供技术支撑
建立了新一代测序数据(包括基因组,RNA-Seq等)和蛋白质质谱数据的分析体系,并应用于基因组的注释及基因表达分析中。与生命医学研究所的其它实验室紧密协作,利用生物信息学的手段和方法对其它实验室产生的高通量数据进行分析,为他们的实验提供新的线索。
1、 肺癌数据库应用系统的构建及肺癌形成过程信号传导途径间的交互作用研究
通过文献挖掘和数据整合,我们构建了肺癌数据库应用系统 (http://www.megabionet.org/bio/hlung)。本系统中从文献中收集了2585个肺癌相关的基因和212个肺癌相关的miRNA,另外,我们还在系统中整合了相关的转录因子的结合位点,每个肺癌基因的SNP信息以及肺癌中与基因调控相关的表观遗传信息。相关文章已发表在Nucleic Acids Research上。
在此基础上,我们进一步在研究肺癌形成过程信号传导途径间的交互作用及机理,并结合其他数据发现与肺癌相关的新基因。
2、 肝癌数据库应用系统的构建及肝癌形成过程信号传导途径间的交互作用研究
通过文献挖掘和数据整合,我们还构建了肝癌数据库应用系统 (http://www.megabionet.org/hcc)。本系统中从文献中收集了2234个肝癌相关的基因和160个肝癌相关的miRNA,另外,我们还在系统中整合了相关的转录因子的结合位点,蛋白质相互作用网络的信息。将每个肝癌相关的蛋白以功能模块的形式展示,便于用户寻找这些蛋白质间的关系。相关文章投递到Cell Research,正在修改。目前,正在进一步研究肝脏疾病(包括肝癌)形成过程中信号传导途径间的交互作用及机理。
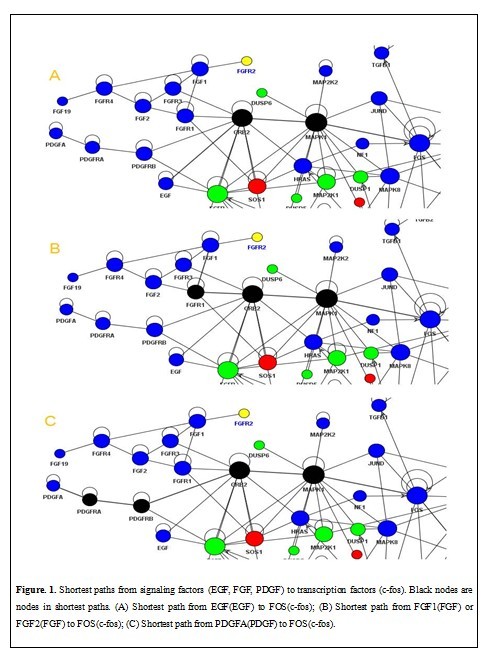
3、 针对芯片数据分析的差异表达基因挑选新方法开发
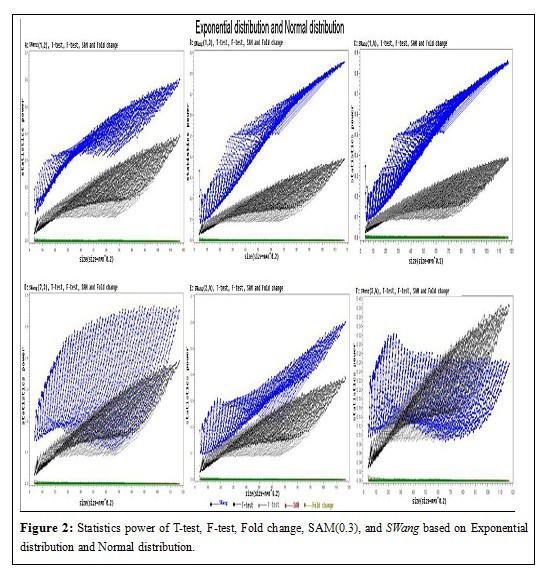
差异表达基因的挑选是基因芯片数据分析的关键一步,所有后续的分析工作都取决于挑选出的差异表达基因。但以往的差异表达基因挑选的统计学方法如T-Test, F-Test, SAM, Fold change + p Value等都是纯统计学方法,没有考虑到生物系统的特异性,故存在着较大的假阳性。我们结合生物系统的特异性,提出了挑选差异表达基因的新统计学方法,实验证明我们的方法可以挑选出其它方法不能发现的差异表达基因。目前,文章已投递到PNAS。
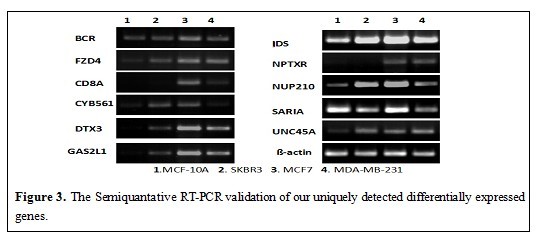
4、 肿瘤诊断特异基因的挑选方法的开发
这些年来,我们一直在参加FDA组织的芯片质控项目(MAQC),即利用芯片数据来提取和发现用于临床疾病检测的特征基因。在此过程中,我们提出了几种不同的方法来挑选疾病相关的特征基因。
(1)开发了减少疾病检测特征基因数量的算法
在不影响疾病检测准确性的前提下,我们的算法可以减少用于疾病检测的特征基因数,从而可以提高临床疾病检测的效率,同时降低成本。
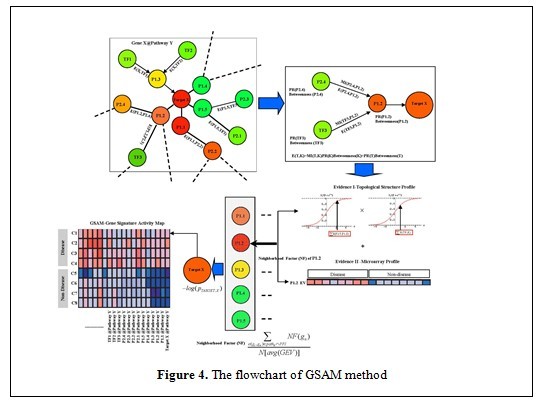
(2)结合网络拓扑学特征,我们将Google的搜寻算法融入到疾病特征基因挑选的过程中,提出了全局的特征基因活性谱(Gene Signature Activity Map, GSAM)方法,应用到几种肿瘤的特征基因的挑选中取得了好的结果。
以上相关的两篇文章已投递到The Pharmacogenomics Journal。另外,我们参与的MAQC项目文章已投递到Nature Biotechnology,正在修改中。
- Chen G, Yin K, Shi LM, Fang Y, Qi Y, Li P, Luo J, He B, Liu MY, Shi TL*. Comparative analysis of human protein-coding and noncoding RNAs between brain and 10 mixed cell lines by RNA-Seq. PLoS One. 2011, 6(11): e28318.
- Chen G, Li RY, Shi LM, Qi JY, Hu PZ, Luo J, Liu MY, Shi TL*. Revealing the missing expressed genes beyond the human reference genome by RNA-Seq. BMC Genomics. 2011, 12(1):590.
- Chen G, Wang C, Shi TL* Overview of available methods for diverse RNA-Seq data analyses. Sci. China Life Sci. 2011, 54(12):1121-1128
- Chen G, Yin KP, Wang C, Shi TL* De novo transcriptome assembly of RNA-Seq reads with different strategies. Sci China Life Sci. 2011, 54(12):1129-1133.
- Zhao C, Shi LM, Tong WD, Shaughnessy JD, Oberthuer A, Pusztai L, Deng YP, Symmans WF, Shi TL*. Maximum predictive power of the microarray-based models for clinical outcomes is limited by correlation between endpoint and gene expression profile. BMC Genomics. 2011, 12(Suppl 5):S3.
- He J, Cui L, Zeng Y, Wang G, Zhou P, Yang Y, Ji L, Zhao Y, Chen J, Shi TL, Zhang P, Wang Z, Chen R, Li X. REGgamma is associated with multiple oncogenic pathways in human cancers. BMC Cancer. 2012, 12(1):75.
- Wang JG, Qiu XJ, Li YH, Shi TL*. A dynamic network of transcription during Arabidopsis thaliana pollen development. BMC Systems Biol. 2011, 5(Suppl 3):S8.
- Chen J, Li T, Wu Y, He L, Zhang L, Shi TL, Yi Z, Liu M, Pang X. Prognostic significance of vascular endothelial growth factor expression in gastric carcinoma: a meta-analysis. J Cancer Res Clin Oncol. 2011, 7(12):1799-812.
- Chang C, Wang JW, Zhao C, Fostel J, Tong WD, Bushel PR, Deng YP, Pusztai L, Symmans WF, Shi TL*. Maximizing biomarker discovery by minimizing gene signatures. BMC Genomics. 2011, 12(Suppl 5):S6.
- Xu F, Zhao C, Li YH, Li J, Shi TL*. Exploring virus relationships based on virus-host protein-protein interaction network. BMC Systems Biol 2011, 5(Suppl 3):S11.
- He B, Zhang H, Shi TL*. A comprehensive analysis of the dynamic biological networks in HCV induced hepatocarcinogenesis. PLoS One 6(4):e18516, 2011.
- (12). Li P, Zang W, Li Y, Xu F, Wang J and Shi TL*. AtPID: the overall hierarchical functional protein interaction network interface and analytic platform for Arabidopsis. Nucleic Acids Res. 2011, 39: D1130-1133.
- (13). Cui J, Liu J, Li Y, Shi TL*. Integrative identification of Arabidopsis mitochondrial proteome and its function exploitation through protein interaction network. PLoS One 2011, 6(1):e16022.


